- 904.71 KB
- 2022-06-16 14:42:13 发布
- 1、本文档共5页,可阅读全部内容。
- 2、本文档内容版权归属内容提供方,所产生的收益全部归内容提供方所有。如果您对本文有版权争议,可选择认领,认领后既往收益都归您。
- 3、本文档由用户上传,本站不保证质量和数量令人满意,可能有诸多瑕疵,付费之前,请仔细先通过免费阅读内容等途径辨别内容交易风险。如存在严重挂羊头卖狗肉之情形,可联系本站下载客服投诉处理。
- 文档侵权举报电话:19940600175。
基因组学与应用生物学,2014年,第33卷,第3期,第695-703页GenomicsandAppliedBiology,2014,Vol.33,No.3,695-703评述与展望ReviewandProgressDNA折纸术的研究进展吴忠钰刘竟然尹俊张峰*内蒙古农业大学生命科学学院,呼和浩特,010018*通讯作者,fengzhang1978@hotmail.com摘要为了帮助广大研究者在短时间内了解DNA折纸术的最新研究现状,掌握DNA折纸术相关软件的应用,本论文将DNA折纸术的相关内容进行了归纳。首先介绍了DNA折纸术的起源与基本原理;然后综述了近几年国内外的研究进展与取得的成果;接下来对DNA折纸术相关软件-Tiamat和Cadnano的应用和功能进行了简单的介绍,并对不同的软件进行了比较;最后对DNA折纸术的应用前景进行了展望。DNA折纸术虽然取得了一定的研究进展,但其未来的发展和应用前景将更广阔。关键词DNA折纸术,自组装,立体结构,Tiamat,CadnanoTheResearchAdvancesinDNAOrigamiWuZhongyuLiuJingranYinJunZhangFeng*CollegeofLifeScience,InnerMongoliaAgriculturalUniversity,Hohhot,010018*Correspondingauthor,fengzhang1978@hotmail.comDOI:10.13417/j.gab.033.000695AbstractTohelptheresearcherslearnaboutthelatestresearchadvancesofDNAorigami,andusetherelatedsoftware"sapplicationsassoonaspossible,thispapersummarisedtherelatedcontentsofDNAorigami.Firstly,weintroducedtheoriginandthebasicprinciplesofDNAorigami.Secondly,wereviewedtheadvancesandresultsofrecentyears.Thirdly,webrieflyintroducedtherelatedsoftware,suchasTiamat,Cadnano,andmadeacomparison.Finally,weoutlookedtheapplicationprospectsofDNAorigami.TheresearchesofDNAorigamihavemadesomeprogresses,butdevelopmentandapplicationinthefuturearemorepromising.KeywordsDNAorigami,DNAself-assembly,Stereostructure,Tiamat,CadnanoDNA折纸术(DNAorigami)是近几年新提出的一种DNA自组装的方法,是DNA纳米技术的一个重要方向,它主要涉及到分子生物学、计算机科学、纳米技术和数学等学科,是一个新型的交叉型科学技术,在新兴的纳米研究领域中具有广阔的应用前景。目前,国内关于该技术的论文或专著还比较少,所以本论文通过查阅近几年的相关文献,综述了该技术的研究进展与成果,并对目前两款热点软件进行了简单介绍与比较,希望能对国内相关领域的研究者有所帮助,使他们能够更好的了解该技术的最新研究现状,从而推动该技术在国内的发展。1DNA折纸术的起源与基本原理1.1DNA折纸术的起源DNA具有结构稳定、自我识别能力强、尺度小、易于操作等优点,一直是科学家们的研究重点。上世纪80年代,Seeman首先提出利用碱基互补配对的原则可将DNA组装成复杂的空间结构(Seeman,1982;SeemanandKallenbach,1983;Seeman,2003),并将这一技术命名为DNA纳米技术。随后,许多科学家投入到这项研究中,并利用这种技术成功的制备出许多功能性的纳米材料(LiaoandSeeman,2004;ShinandPierce,2004;Seeman,2005;Tianetal.,2005)。DNA折纸术这一全新的名词最早于2006年由Rothemund(2006)提出,他将特殊设计的200多条订书钉链(每条长约为32个碱基)与DNA长链(M13mp18)进行碱基配对,成功的折叠出长方形、三角形、五角星与笑脸等图形(图1),其文章以封面论文的形式发表于Nature杂志上。另外,Rothemund以DNA折叠得到的有缝矩形结构为基板,利用DNA基金项目:本研究由国家自然科学基金项目(21171086)、国家自然科学基金项目(81160213)、内蒙古自治区草原英才工程项目(108-108038)和内蒙古自治区自然科学基金项目(2013MS1121)共同资助
DNA折纸术的研究进展TheResearchAdvancesinDNAOrigami6962近几年国内外的研究进展与研究成果图1首次得到的DNA折纸图形(Rothemund,2006)Figure1ThefirstDNAorigamiimages(Rothemund,2006)自2006年Rothemund首先提出了DNA折纸术这一新名词后,许多科学家也先后纷纷投入到这项研究中,成功的构造出许多二维、三维的结构或图形,并取得了瞩目的成果。DNA折纸术的发展首先体现在图形构建方面。Andersen等(2008)利用DNA折纸术成功的设计出奥胡斯大学海豚结构的校徽,并通过调整海豚尾巴处Crossover的数目来控制海豚尾巴的活动状况,从而得到了形状各异的图形,这是第一张有孔的非对称图形(图2A)。次年,他们又以M13mp18作为脚手架链设计出一个3D盒子(图2B),并且通过链的置换反应可以实现盒子盖的开关(Andersenetal.,2009)。3个月后,该实验室的Dietz等(2009)又设计出各式各样的扭转图形,角度能精确到3度左右。时间相隔不久,Ke等(2009)设计出一个空心的四面体立体结构(图2c)。随后,Komiyama等(2009)将Shih的方法进行了改善,选择性的加入跨面订书钉链,也成功的得到了一个立方体结构。折纸术的可寻址性,将特定位点的订书钉链修饰成带有哑铃状的碱基序列,成功的设计出字母、雪花、美洲地图等图案,因为哑铃状的结构比其他的订书钉链高,在AFM下即可观察到人为设计的图案。值得指出的是这篇文章的作者只有Rothemund一人,但附件却有一百页之多,里面对设计过程中涉及到的许多细节问题进行了详细的描述和解答,为后来的研究者提高了宝贵的参考资料。1.2DNA折纸术的基本原理DNA折纸术的基本原理是将一条较长的DNA单链(脚手架链,scaffoldstrand,通常用的是基因组DNA,如M13mp18,P7308,P7560等)与一系列经过特殊设计的短DNA片段(订书钉链,staplestrand)通过碱基配对原则进行互补,按照人的意愿可控地构造出理想的高度复杂的纳米结构或图案。最终得到的图案或结构大小、形状取决于特殊的订书钉链。与传统的DNA自组装相比,DNA折纸术更容易构造出高度复杂、结构稳定的纳米级结构,并具有可寻址性,是DNA自组装与DNA纳米技术领域的一个重大进展。图2DNA折纸图形Figure2ImagesofDNAorigami当DNA折纸术取得突飞猛进的发展时,科学家们碰见了一个难题。之前研究所用的脚手架链大部分是M13mp18,它是由7249个核苷酸组成,所以设计得到的图形大小一直受限制,因而扩大DNA折纸术图形的规模成为许多科学家的奋斗目标。Zhao等(2010)在AngewandteChemie发表的一
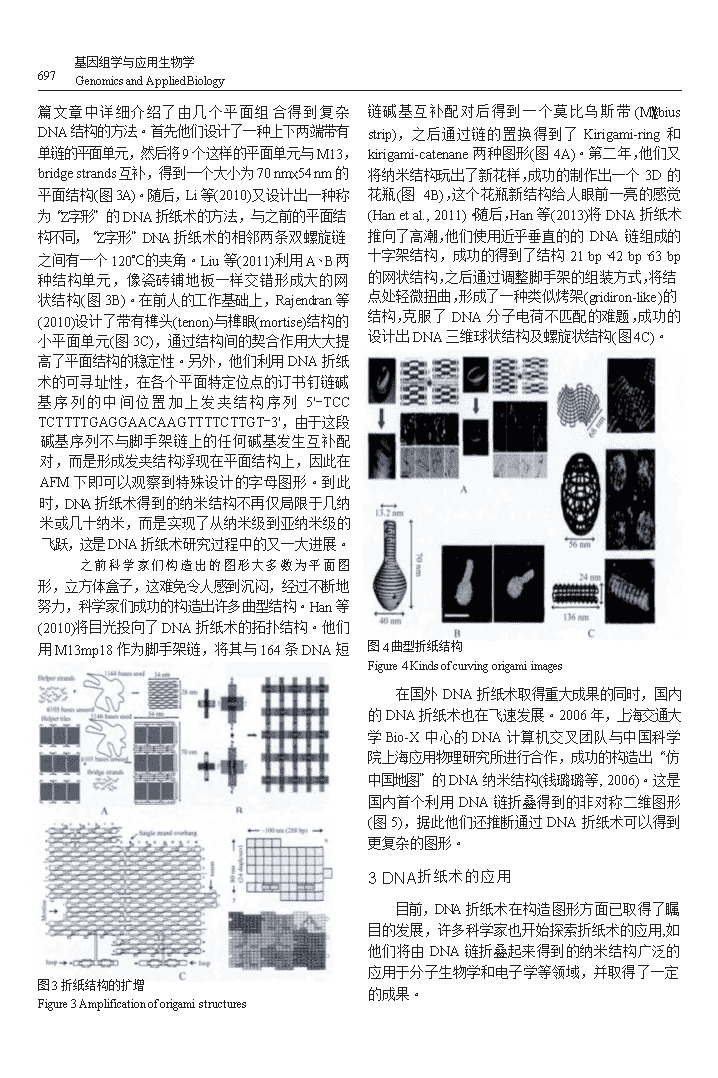
基因组学与应用生物学697GenomicsandAppliedBiology篇文章中详细介绍了由几个平面组合得到复杂DNA结构的方法。首先他们设计了一种上下两端带有单链的平面单元,然后将9个这样的平面单元与M13,bridgestrands互补,得到一个大小为70nm×54nm的平面结构(图3A)。随后,Li等(2010)又设计出一种称为“Z字形”的DNA折纸术的方法,与之前的平面结构不同,“Z字形”DNA折纸术的相邻两条双螺旋链之间有一个120℃的夹角。Liu等(2011)利用A、B两种结构单元,像瓷砖铺地板一样交错形成大的网状结构(图3B)。在前人的工作基础上,Rajendran等(2010)设计了带有榫头(tenon)与榫眼(mortise)结构的小平面单元(图3C),通过结构间的契合作用大大提高了平面结构的稳定性。另外,他们利用DNA折纸术的可寻址性,在各个平面特定位点的订书钉链碱基序列的中间位置加上发夹结构序列5"-TCCTCTTTTGAGGAACAAGTTTTCTTGT-3",由于这段碱基序列不与脚手架链上的任何碱基发生互补配对,而是形成发夹结构浮现在平面结构上,因此在AFM下即可以观察到特殊设计的字母图形。到此时,DNA折纸术得到的纳米结构不再仅局限于几纳米或几十纳米,而是实现了从纳米级到亚纳米级的飞跃,这是DNA折纸术研究过程中的又一大进展。之前科学家们构造出的图形大多数为平面图形,立方体盒子,这难免令人感到沉闷,经过不断地努力,科学家们成功的构造出许多曲型结构。Han等(2010)将目光投向了DNA折纸术的拓扑结构。他们用M13mp18作为脚手架链,将其与164条DNA短图3折纸结构的扩增Figure3Amplificationoforigamistructures链碱基互补配对后得到一个莫比乌斯带(M觟biusstrip),之后通过链的置换得到了Kirigami-ring和kirigami-catenane两种图形(图4A)。第二年,他们又将纳米结构玩出了新花样,成功的制作出一个3D的花瓶(图4B),这个花瓶新结构给人眼前一亮的感觉(Hanetal.,2011)。随后,Han等(2013)将DNA折纸术推向了高潮,他们使用近乎垂直的的DNA链组成的十字架结构,成功的得到了结构21bp、42bp、63bp的网状结构,之后通过调整脚手架的组装方式,将结点处轻微扭曲,形成了一种类似烤架(gridiron-like)的结构,克服了DNA分子电荷不匹配的难题,成功的设计出DNA三维球状结构及螺旋状结构(图4C)。图4曲型折纸结构Figure4Kindsofcurvingorigamiimages在国外DNA折纸术取得重大成果的同时,国内的DNA折纸术也在飞速发展。2006年,上海交通大学Bio-X中心的DNA计算机交叉团队与中国科学院上海应用物理研究所进行合作,成功的构造出“仿中国地图”的DNA纳米结构(钱璐璐等,2006)。这是国内首个利用DNA链折叠得到的非对称二维图形(图5),据此他们还推断通过DNA折纸术可以得到更复杂的图形。3DNA折纸术的应用目前,DNA折纸术在构造图形方面已取得了瞩目的发展,许多科学家也开始探索折纸术的应用,如他们将由DNA链折叠起来得到的纳米结构广泛的应用于分子生物学和电子学等领域,并取得了一定的成果。
DNA折纸术的研究进展TheResearchAdvancesinDNAOrigami698图5中国地图的原子力显微镜图像(钱璐璐等,2006)Figure5AFMimageofthemapofchina(Qianetal.,2006)3.1利用折纸术组装纳米颗粒DNA具有易被化学修饰的特点,被修饰后的DNA分子可与纳米金、量子点等生物分子“共价”螯合,因此,根据DNA折纸术的可寻址性,可实现纳米金、量子点的纳米颗粒的有序排列。在纳米颗粒的排布方面做得最出色的应该要属Yan实验室。2007年,他们将标记着生物素(biotin)的DNA通过自组装折叠成纳米管(图6),将制备的纳米管结合上链霉亲和素(STV)-量子点(QD)复合物,利用自下而上和自上而下的工艺,最终得到了介观尺度排列的量子点(Linetal.,2007)。次年,他们在DNA平面上又实现了量子点的整齐排布(Sharmaetal.,2008)。将A、B、C、D四种结构的瓦片(tile)组合起来得到一个平面结构,其中A型瓦片的特定位置修饰有生物素,然后与其包被量子点的链霉亲和素结合,从而实现了量子点的整齐排布(图7)。除经常在DNA结构上对量子点进行排列外,金图6链霉亲和素-量子点整齐排列的纳米管(Linetal.,2007)Figure6DNAnanotubeswithstreptavidin-quantumdotconju-gatearrays(Linetal.,2007)图7含有量子点序列的DNA瓷砖自组装示意图(Sharmaetal.,2008)Figure7ProcessofDNA-tile-directedself-assemblyofQDarrays(Sharmaetal.,2008)颗粒由于可通过静电吸附与氨基进行结合,也可与巯基形成稳定的Au-S键,所以很容易与DNA结合到一起,因此也是一种常被用来进行排布的模型纳米颗粒。之前,许多科学家对纳米金颗粒的排布进行过实验,但是产率非常低(Xiaoetal.,2002;Leetal.,2004;Dengetal.,2005;Pintoetal.,2005;Sharmaetal.,2006;Zhangetal.,2006)。Sharma等(2008)用酯化的硫辛酸代替了之前用来修饰DNA的硫醇,酯化的硫酸锌具有两个能与金颗粒结合的硫键(图8),比之前所用的硫醇多了一个硫键,这样大大的提高了结合效率,使两个金颗粒同时结合到矩形折纸基板的效率达到了90%左右。随后,Ding等(2009)将3个不同直径的纳米金颗粒分别结合到3条DNA链上,然后让它们与另外5条DNA链互补配对,形成TXtile结构,在扫描电子显微镜下成功的观察到了3个等间距排列的大小的金颗粒。由上述研究成果可知,利用DNA分子的易修饰图8结合两个金颗粒的DNA折纸基板(Sharmaetal.,2008)Figure8DNAorigamicombinedwithtwogoldnanoparticles(Sharmaetal.,2008)
基因组学与应用生物学699GenomicsandAppliedBiology性与折纸术的可寻址性,能够精确地排布金颗粒与量子点等纳米颗粒,这将在光电子器件、显微镜学、检测器和共振器方面有很大的应用价值。3.2利用折纸术组装蛋白质分子蛋白质作为生命活动的体现者一直是科学家的研究热点,在利用DNA折纸术对纳米金、量子点等纳米颗粒进行可控排列的同时,他们也尝试着在DNA纳米结构上精确地排布蛋白质分子,并期待着将蛋白质类药物连接上去,从而利用DNA的特异性,将药物精确地传输到病变部位,达到治疗疾病的目的。目前,科学家通过核酸杂交(Wilneretal.,2009)、适配子特异结合(Liuetal.,2005;Chhabraetal.,2007;Rinkeretal.,2008)、生物素-链霉亲和素反应(Cohenetal.,2007;Cohenetal.,2008;Kuzuyaetal.,2009;Numajirietal.,2010;Voigtetal.,2010)、抗体-抗原识别(Heetal.,2006;Williamsetal.,2007)等途径,已成功地将蛋白质结合到了DNA纳米结构上。Williams等(2007)通过抗体-抗原识别的方法,在由四种瓦片组成的DNA基板上特异性地将蛋白质进行了排布。Rinker等(2008)通过控制DNA基板上的适配子间的距离,使同一个蛋白成功的结合到两个适配子上面(图9),首次实现了蛋白质在DNA基板上的多价结合。Anton等(2009)通过先将蛋白质连接到DNA上再将DNA进行组装折叠和先让DNA折叠组装成纳米结构再结合上蛋白质这两种方法进行生物素-链霉亲和素的排布,实验发现这两种方图9DNA折纸基板上蛋白质的多价结合(Rinkeretal.,2008)Figure9ThemultivalentbindingproteinonDNAorigami(Rinkeretal.,2008)法都能将蛋白质结合到DNA纳米结构上,并且得到的设计图案的成功率较高。同年,Kuzuya等(2009)将折纸结构上设计有规则排布的纳米孔结构,并在每个纳米孔的外壁上修饰上两个生物素分子,通过生物素与链霉亲和素的电子吸附作用以及孔的限定作用,能将链霉亲和素稳定的结合在折纸结构上。3.3利用折纸术设计芯片DNA折叠后得到的折纸结构除能结合纳米颗粒、蛋白等生物分子外,还能制作各种芯片。例如制作DNA芯片时,可将某一位置的订书钉链延长当作探针;另外,在订书钉链上连接上抗原,即得到了抗体探针。Ke等(2008)制作出第一张具有探测RNA序列功能的折纸术芯片,他们是以长方形的折纸结构为基板,将具体位点的订书链末端延长,得到伸出的V字型的探针,这些探针能与溶液中的RNA杂交,在长方形的折纸结构平台上形成凸起,用AFM观察很容易成像(图10)。这种芯片具有不用标记目标链、检测灵敏度较高的优点,但是得到的折纸图形不同位置的探针杂交效率不相同,即有位置效应,并且芯片的扩展性不强。之后,上海交通大学对Ke的方法进行了改进,将V字型探针改成了一字型型探针,并引入了biotin-STV反应,使信号的强度得到增强,成功的克服了位置效应,他们的这种方法不需要做索引标记,并且增加了探针的可寻址性。Subramanian等(2011)设计出能探测单核苷酸多态性(singlenucleotidepolymorphism,SNP)的芯片,利用原子力显微镜可以观察到分子水平的SNP信号(图11)。3.4利用折纸术研究单分子反应机理2010年,Voigt等(2010)另辟蹊径,发现了DNA图10第一张RNA芯片(Keetal.,2008)Figure10ImageoffirstRNAchip(Keetal.,2008)
DNA折纸术的研究进展TheResearchAdvancesinDNAOrigami700图11SNP的原子力显微镜图像(Subramanianetal.,2011)Figure11AFMimageofthesinglenucleotidepolymorphisms(Subramanianetal.,2011)折纸术的又一大用处,即可用于单分子的检测研究。他们在矩形DNA折纸结构上修饰几个特定的化学基团,然后加入一系列的化合物,促进生物素与链霉亲和素间化学键的合成与断裂,在原子力显微镜下观察物质的合成与分解(图12)。与传统的方法相比,DNA折纸术能够精确地定位每个分子的空间位置,在分子水平上研究物质的反应机理;而传统的方法只是在宏观上表征物质的反应。另外,Nangreave等(2009)以DNA折纸结构为模板对多价态反应进行了系统性研究。他们通过改变DNA黏性末端的排布,利用荧光共振能量转移技术实时监测DNA结构组装过程中的温度和能量的变化,发现DNA黏性末端的数量与位置对DNA折纸结构的稳定性起很重要的作用,这为DNA纳米结构稳定性的研究奠定了坚实的基础。3.5折纸术在其它方面的应用科学家们利用DNA折纸术的可寻址性与分子间化学键的合成与断裂,成功地使某些特殊结构能图12单分子反应检测示意图(Voigtetal.,2010)Figure12ThediagramimageofSingle-moleculechemicalreac-tions(Voigtetal.,2010)够按人为设计的轨道“行走”,这为在纳米尺寸上组装电子和光学器件提供了有效途径,为多功能材料的构建奠定了坚实的基础。Gu等(2010)设计出能收集金纳米颗粒的“Walker”,这个类似于三角形的结构有三条腿,能够选择性地结合到DNA基板上不同位点的核酸单链,当结合位点完全正确时,就可以将纳米金颗粒结合上去(图13)。无独有偶,Lund(2010)设计出一个以链霉亲和素作为身体、脱氧核酸酶为腿的行走者,并形象的称它为爬行蜘蛛(spider),它能按照人为设定的轨道行走(图14)。图13能结合纳米金颗粒的“Walker”(Guetal.,2010)Figure13Thewalkercanpickupagoldnanoparticle(Guetal.,2010)图14按特定核酸轨道行走的链霉亲和素Spider(Lundetal.,2010)Figure14Thestreptavidinspiderwalkedalonganucleicacidtrack(Lundetal.,2010)4DNA折纸术的相关软件在DNA折纸术飞速发展的同时,人们也编写了许多款创建DNA折纸术结构的软件,但是使用率最高的应属Tiamat和Cadnano,但是这两款软件也各有优缺点。YAN实验室利用Tiamat软件成功的设计出许多平面图形,例如21bp、42bp等平面结构,这个软件在创建DNA链的时候,软件能自动弹出一个对话窗口,可直接输入链的碱基数目,设计出来的结构,能更好的体现出DNA螺旋的空间结构,而且人为地设定某个碱基的类型更为方便,但是由于窗口的界面有限,如果结构太大,将不能呈现在视野内,只能通过缩放和来回拖放定位到某一区域。相对于
基因组学与应用生物学701GenomicsandAppliedBiologyTiamat,Cadnano软件设计在设计三维图形更直观些,它有honeycomblattice和squarelattice两种样式,并且脚手架链可以直接手动设定,不用直接输入,这比Tiamat方便的多。Cadnano与MAYA软件结合到一起,使其功能更为强大,但是利用Cadnano设计某一条DNA双链时,其长度不如在Tiamat软件的对话框中直接输入方便,它需要拖动鼠标手动控制,并且双链间的空间结构也不如Tiamat中的直观。通过两款软件的实际运用,个人觉得Tiamat用来设计平面结构方便些,而Cadnano适合用来设计立体结构。图15是分别利用这两款软件设计出来的图形。图15利用Tiamat和Cadnano软件设计的结构Figure15StructuresdesignedbytheTiamat,CadnanoSoftwarerespectively5展望DNA折纸术作为一个新兴技术,在短短的几年内已经取得了瞩目的成就,而DNA以其独特的稳定性、识别性及其自组装的优势,正被广泛的应用于电子和生物学领域,设计得到的纳米结构为化学与生物分子相互作用的研究提供了平台(Belletal.,2011;Weietal.,2012),这吸引着越来越多的科学家投入到这个领域中。目前,虽然利用DNA折纸术的可寻址性与方向的可控性成功得到了DNA芯片,实现了纳米、蛋白质、纳米金颗粒等有序排布,但是随着研究的深入,我们也发现了许多问题,例如,折叠过程中的热力学机制尚未明确,许多研究者期待的药物传递与定点投放(Douglasetal.,2012)等研究也不尽人意,并且利用DNA折纸术设计得到的许多成果也局限于试验阶段,还未真正的投入到生活应用中,DNA折纸术创建的模型也比较单调,非对称的立体模型少之又少,而且设计模型的几款软件也是美中存在着不足,所以DNA折纸术仍需科学家们的不断研究与探索,仍有很大的发展空间及应用前景。作者贡献吴忠钰和刘竟然阅读文献,整理图片并撰写论文;张峰和尹俊负责论文构思,设计逻辑结构,编写、审阅论文并提出修改意见。致谢感谢国家自然科学基金项目(21171086)、国家自然科学基金项目(81160213)、内蒙古自治区草原英才工程项目(108-108038)和内蒙古自治区自然科学基金项目(2013MS1121)的资助。参考文献AndersenE.S.,DongM.,NielsenM.M.,andJahnK.,Lind-ThomsenA.,MamdouhW.,GothelfK.V.,Besenbach-erF.,andKjemsJ.,2008,DNAorigamidesignofdol-phin-shapedstructureswithflexibletails,ACSNano,2(6):1213-1218AndersenE.S.,DongM.,NielsenM.M.,JahnK.,SubramaniR.,MamdouhW.,GolasM.M.,SanderB.,StarkH.,OliveiraC.L.P.,PedersenJ.S.,BirkedalV.,BesenbacherF.,GothelfK.V.,andKjemsJ.,2009,Self-assemblyofananoscaleDNAboxwithacontrollablelid,Nature,459(7243):73-76AntonK.,KimmoT.L.,andT觟rm覿P.,2009,DNAorigamiasananoscaletemplateforproteinassembly,Nanotechnology,20(23):235305BellN.A.,EngstC.R.,AblayM.,DivitiniG.,DucatiC.,LiedlT.,andKeyserU.F.,2011,DNAorigaminanopores,NanoLet-ters,12(1):512-517ChhabraR.,SharmaJ.,KeY.,LiuY.,RinkerS.,LindsayS.,andYanH.,2007,Spatiallyaddressablemultiproteinnanoarraystemplatedbyaptamer-taggedDNAnanoarchitectures,JournaloftheAmericanChemicalSociety,129(34):10304-10305CohenJ.D.,SadowskiJ.P.,andDervanP.B.,2007,AddressingsinglemoleculesonDNAnanostructures,AngewandteChemie,119(42):8102-8105CohenJ.D.,SadowskiJ.P.,andDervanP.B.,2008,ProgrammingmultipleproteinpatternsonasingleDNAnanostructure,JournaloftheAmericanChemicalSociety,130(2):402-403DengZ.,TianY.,LeeS.H.,RibbeA.E.,andMaoC.,2005,DNA-encodedself-assemblyofgoldnanoparticlesintoone-dimensionalarrays,AngewandteChemie,117(23):3648-3651DietzH.,DouglasS.M.,andShihW.M.,2009,FoldingDNAintotwistedandcurvednanoscaleshapes,Science,325(5941):725-730DingB.,CabriniS.,ZuckermannR.N.,andBokorJ.,2009,DNAdirectedassemblyofnanoparticlelinearstructurefornanophotonics,JournalofVacuumScience&TechnologyB,27(1):184-187DouglasS.M.,BacheletI.,andChurchG.M.,2012,Alogic-gatednanorobotfortargetedtransportofmolecularpayloads,Sci-ence,335(6070):831-834GuH.,ChaoJ.,XiaoS.J.,andSeemanN.C.,2010,Aproximity-basedprogrammableDNAnanoscaleassemblyline,Nature,
DNA折纸术的研究进展TheResearchAdvancesinDNAOrigami702465(7295):202-205HanD.,PalS.,LiuY.,andYanH.,2010,FoldingandcuttingDNAintoreconfigurabletopologicalnanostructures,NatureNanotechnology,5(10):712-717HanD.,PalS.,NangreaveJ.,DengZ.,LiuY.,andYanH.,2011,DNAorigamiwithcomplexcurvaturesinthree-dimensionalspace,Science,332(6027):342-346HanD.,PalS.,YangY.,JiangS.,NangreaveJ.,LiuY.,andYanH.,2013,DNAgridironnanostructuresbasedonfour-armjunctions,Science,339(6126):1412-1415HeY.,TianY.,RibbeA.E.,andMaoC.,2006,Antibodynanoar-rayswithapitchof20nanometers,JournaloftheAmericanChemicalSociety,128(39):12664-12665KeY.,LindsayS.,ChangY.,LiuY.,andYanH.,2008,Self-as-sembledwater-solublenucleicacidprobetilesforlabel-freeRNAhybridizationassays,Science,319(5860):180-183KeY.,SharmaJ.,LiuM.,JahnK.,LiuY.,andYanH.,2009,ScaffoldedDNAorigamiofaDNAtetrahedronmolecularcontainer,NanoLetters,9(6):2445-2447KuzuyaA.,andKomiyamaM.,2009,Designandconstructionofabox-shaped3D-DNAorigami,ChemicalCommunications,28:4182-4184KuzuyaA.,KimuraM.,NumajiriK.,KoshiN.,OhnishiT.,Oka-daF.,andKomiyamaM.,2009,Preciselyprogrammedandrobust2Dstreptavidinnanoarraysbyusingperiodicalnanometer-scalewellsembeddedinDNAorigamiassembly,Chem.Bio.Chem.,10(11):1811-1815LeJ.D.,PintoY.,SeemanN.C.,Musier-ForsythK.,TatonT.A.,andKiehlR.A.,2004,DNA-templatedself-assemblyofmetallicnanocomponentarraysonasurface,NanoLetters,4(12):2343-2347LiZ.,LiuM.,WangL.,NangreaveJ.,YanH.,andLiuY.,2010,MolecularbehaviorofDNAorigamiinhigher-orderself-as-sembly,JournaloftheAmericanChemicalSociety,132(38):13545-13552LiaoS.,andSeemanN.C.,2004,TranslationofDNAsignalsintopo-lymerassemblyinstructions,Science,306(5704):2072-2074LinC.,KeY.,LiuY.,MertigM.,GuJ.,andYanH.,2007,Func-tionalDNAnanotubearrays:Bottom-upmeetstop-down,AngewandteChemie,119(32):6201-6204LiuW.,ZhongH.,WangR.,andSeemanN.C.,2011,Crystallinetwo-dimensionalDNA-origamiarrays,AngewandteChemie,123(1):278-281LiuY.,LinC.,LiH.,andYanH.,2005,Aptamer-directedself-assemblyofproteinarraysonaDNAnanostructure,AngewandteChemie,117(28):4407-4412LundK.,ManzoA.J.,DabbyN.,MichelottiN.,Johnson-BuckA.,NangreaveJ.,TaylorS.,PeiR.,StojanovicM.N.,WalterN.G.,WinfreeE.,andYanH.,2010,Molecularrobotsguidedbyprescriptivelandscapes,Nature,465(7295):206-210NangreaveJ.,YanH.,andLiuY.,2009,Studiesofthermalsta-bilityofmultivalentDNAhybridizationinananostructuredsystem,BiophysicalJournal,97(2):563-571NumajiriK.,YamazakiT.,KimuraM.,KuzuyaA.,andKomiyamaM.,2010,DiscreteandactiveenzymenanoarraysonDNAorigamiscaffoldspurifiedbyaffinitytagseparation,JournaloftheAmericanChemicalSociety,132(29):9937-9939PintoY.Y.,LeJ.D.,SeemanN.C.,Musier-ForsythK.,TatonT.A.,andKiehlR.A.,2005,Sequence-encodedself-assemblyofmultiple-nanocomponentarraysby2DDNAscaffolding,NanoLetters,5(12):2399-2402QianL.L.,WangY.,ZhangZ.,ZhaoJ.,PanD.,ZhangY.,LiuQ.,FanC.H.,HuJ.,andHeL.,2006,DNAnanostructurescopymapofChina,KexueTongbao(ScienceBulletin),51(24):2860-2863(钱璐璐,汪颖,张钊,赵健,潘敦,张益,刘强,樊春海,胡钧,贺林,2006,DNA纳米结构仿中国地图,科学通报,51(24):2860-2863)RajendranA.,EndoM.,KatsudaY.,HidakaK.,andSugiyamaH.,2010,Programmedtwo-dimensionalself-assemblyofmulti-pleDNAorigamijigsawpieces,ACSNano,5(1):665-671RinkerS.,KeY.,LiuY.,ChhabraR.,andYanH.,2008,Self-as-sembledDNAnanostructuresfordistance-dependentmulti-valentligand-proteinbinding,NatureNanotechnology,3(7):418-422RothemundP.W.,2006,FoldingDNAtocreatenanoscaleshapesandpatterns,Nature,440(7082):297-302SeemanN.C.,1982,Nucleicacidjunctionsandlattices,JournalofTheoreticalBiology,99(2):237-247SeemanN.C.,2003,DNAinamaterialworld,Nature,421(6921):427-431SeemanN.C.,2005,Fromgenestomachines:DNAnanome-chanicaldevices,TrendsinBiochemicalSciences,30(3):119-125SeemanN.C.,andKallenbachN.R.,1983,Designofimmobilenucleicacidjunctions,BiophysicalJournal,44(2):201-209SharmaJ.,ChhabraR.,AndersenC.S.,GothelfK.V.,YanH.,andLiuY.,2008,Towardreliablegoldnanoparticlepattern-ingonself-assembledDNAnanoscaffold,JournaloftheAmericanChemicalSociety,130(25):7820-7821SharmaJ.,ChhabraR.,LiuY.,KeY.,andYanH.,2006,DNA-templatedself-assemblyoftwo-dimensionalandperi-odicalgoldnanoparticlearrays,AngewandteChemieInter-nationalEdition,45(5):730-735SharmaJ.,KeY.,LinC.,ChhabraR.,WangQ.,NangreaveJ.,LiuY.,andYanH.,2008,DNA-tile-directedself-assemblyofquantumdotsintotwo-dimensionalnanopatterns,Ange-wandteChemieInternationalEdition,47(28):5157-5159ShinJ.S.,andPierceN.A.,2004,AsyntheticDNAwalkerformoleculartransport,JournaloftheAmericanChemicalSo-ciety,126(35):10834-10835SubramanianH.K.,ChakrabortyB.,ShaR.,andSeemanN.C.,2011,Thelabel-freeunambiguousdetectionandsymbolicdisplayofsinglenucleotidepolymorphismsonDNAorigami,NanoLetters,11(2):910-913TianY.,HeY.,ChenY.,YinP.,andMaoC.,2005,ADNAzymethatwalksprocessivelyandautonomouslyalongaOne-di-
基因组学与应用生物学703GenomicsandAppliedBiologymensionaltrack,AngewandteChemieInternationalEdition,44(28):4355-4358VoigtN.V.,T覬rringT.,RotaruA.,JacobsenM.F.,Ravnsb覸kJ.B.,SubramaniR.,MamdouhW.,KjemsJ.,MokhirA.,Besen-bacherF.,andGothelfK.V.,2010,Single-moleculechemicalreactionsonDNAorigami,NatureNanotechnology,5(3):200-203WeiR.,MartinT.G.,RantU.,andDietzH.,2012,DNAorigamigatekeepersforsolid-statenanopores,AngewandteChemie,124(20):4948-4951WilliamsB.A.,LundK.,LiuY.,YanH.,andChaputJ.C.,2007,Self-assembledpeptidenanoarrays:Anapproachtostudyingprotein-proteininteractions,AngewandteChemie,119(17):3111-3114WilnerO.I.,WeizmannY.,GillR.,LioubashevskiO.,FreemanR.,andWillnerI.,2009,EnzymecascadesactivatedontopologicallyprogrammedDNAscaffolds,NatureNan-otechnology,4(4):249-254XiaoS.,LiuF.,RosenA.E.,HainfeldJ.F.,SeemanN.C.,Musier-ForsythK.,andKiehlR.A.,2002,SelfassemblyofmetallicnanoparticlearraysbyDNAscaffolding,JournalofNanoparticleResearch,4(4):313-317ZhangJ.,LiuY.,KeY.,andYanH.,2006,Periodicsquare-likegoldnanoparticlearraystemplatedbyself-assembled2DDNAnanogridsonasurface,NanoLetters,6(2):248-251ZhaoZ.,YanH.,andLiuY.,2010,AroutetoscaleupDNAorigamiusingDNAtilesasfoldingstaples,AngewandteChemie,122(8):1456-1459